-
カテゴリ別
-
場所
-
最新のお知らせ 年間スケジュール(就職対策講座等) OB・OG情報 システムの利用方法 学内合同企業説明会(「九州大学生のための業界・企業研究」) 学内企業研究セミナー / 個別企業説明会 公務員・教員等採用情報 就職相談 就職情報室 キャリア教育 低年次学年向け情報 インターンシップ等(学生向け) TOEIC対策プログラム 博士人材のための就職支援(学生向け) 外国人留学生のための就職支援 未内定の学生及び既卒者(卒業後3年以内)の皆様へ 障害のある学生のための就職支援 部局独自の就職支援 東京・大阪・博多駅オフィスの利用 各種情報サイト 過去の就職状況 採用選考に関する指針 学内合同企業説明会 博士人材のための企業説明会(企業向け) 本学へのご訪問 求人情報ご提供 OB・OG名簿ご提供 インターンシップ等(企業向け) 外国人留学生の採用 採用選考に関する指針 就職担当 よくあるご質問
Research Results 研究成果
タンパク質の大規模精密定量法の開発に成功 -がん研究等への応用に期待-
2016.12.27研究成果Life & HealthPhysics & Chemistry
九州大学生体防御医学研究所の中山敬一主幹教授と松本雅記准教授の研究グループは、網羅的なヒトタンパク質ライブラリー(in vitro proteome)と高感度な定量プロテオミクスの手法であるMRM法を組み合わせることで、あらゆるタンパク質の存在量を正確に測定できる新規テクノロジー『iMPAQT(in vitro proteome-assisted MRM for protein absolute quantification)法』の開発に成功しました。このiMPAQT法を用いて、正常細胞およびがん細胞において1000種類近くある代謝酵素タンパク質の存在量を全て計測することで、がん細胞に特徴的な変化である代謝経路再編成の全体像を明らかにしました(図1)。
様々な生命現象の理解や各種疾患の原因解明および診断法開発に向けて、生命現象の直接的な担い手であるタンパク質の存在量を大規模かつ正確に測定することが求められています。しかしながら、現在普及している従来の解析法では定量精度や再現性、および分析速度が不十分でした。一方、タンパク質の精密定量が可能なMRM法は従来法の欠点を解消できる手法として注目されていますが、MRM測定前に高感度ペプチド(プロテオティピックペプチド:PTP)の選定や測定条件最適化などの手間を要することから普及が遅れていました。この度、本研究グループは、網羅的な組み換えタンパク質リソース(18,000以上のタンパク質)を利用することでMRM法に必要な事前情報および内部標準ペプチドを網羅的に取得し、これを用いて容易に多数のタンパク質の絶対定量が実施可能な新規解析基盤iMPAQT法の構築に成功しました。このiMPAQT法で正常細胞とがん細胞における代謝酵素タンパク質の存在量を比較したところ、正常細胞では異化代謝と呼ばれる経路が主であるのに対し、がん細胞ではむしろ同化代謝が盛んであることがわかり、がん細胞は細胞増殖に有利な代謝状態へ変化していることが明らかとなりました。今後は、がん細胞特有の弱点を見つけるなどの治療標的の探索を進めるとともに、タンパク質発現変化に基づく各種疾患の診断法や治療効果モニタリング法の開発が期待されます。
本研究成果は、2016年12月26日(月)午後4時(英国時間)に英国科学雑誌「Nature Methods」で公開されました。
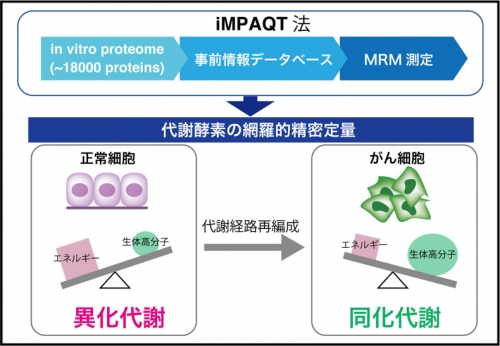
図1:新規プロテオーム解析法iMPAQTによるがん代謝全体像解明
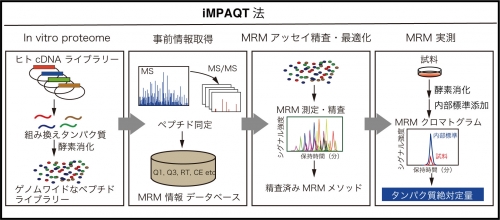
図2:iMPAQT法の概要
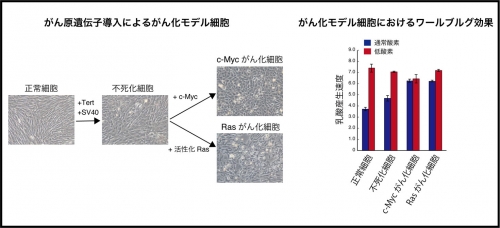
図3:がん原遺伝子導入による人工がん化は代謝変化を引き起こす
研究者からひとこと
全てのタンパク質の存在量を正確に計測できるiMPAQT法の開発によって、がんにおける変化が総合的に追跡できるようになりました。今まで「点」の研究だったものが「面」の研究に広がりを持つようになり、がんの弱点が明らかになりました。将来的にはこの知見ががんに対する薬剤の開発に役立つものと考えています。
- 本研究についての詳細は こちら
論文情報
A large-scale targeted proteomics assay resource based on an in vitro human proteome ,Nature Methods,10.1038/nmeth.4116